PHYTOSOL-2
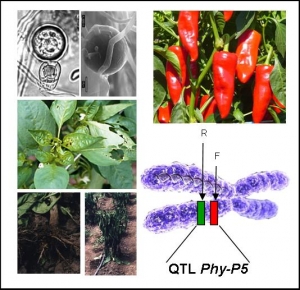
Validation fonctionnelle de gènes candidats pour un QTL à spectre large agissant sur la résistance aux Phytophthora chez les Solanacées
Projet ANR - Réseau de Génomique Végétale - Génoplante 2010
Coordinateur du projet :
Dr. Véronique Lefebvre
INRA-UR1052-GAFL
Caractérisation Fonctionnelle des interactions Plantes-Bioagresseurs
BP94 - 84140 Montfavet - France
Email: veronique.lefebvre@avignon.inra.fr
Résumé :
Les Phytophthora sont les parasites les plus dévastateurs des Dicotylédones. La lutte chimique a été interdite par la commission européenne. Les résistances monogéniques sont fréquemment contournées. Bien que les résistances polygéniques aient été démontrées plus durables, leurs bases moléculaires demeurent inconnues.
Nous avons pour objectif de déterminer la nature moléculaire d’un QTL impliqué dans la résistance à plusieurs espèces de Phytophthora chez les Solanacées. Ce QTL, Phy-P5, confère un niveau élevé de résistance à plusieurs isolats de P. capsici chez le piment et montre une position colinéaire avec des QTL de résistance à P. infestans chez la tomate et la pomme de terre. L’analyse génétique du locus Phy-P5 a permis d’identifier deux QTL finement liés dans 0.57 cM. Deux clones BAC contenant chacun un des deux gènes sous-jacents à Phy-P5 seront séquencés, annotés, et ancrés à la carte génétique en 2007, afin de sélectionner les gènes candidats.
Les objectifs du projet PHYTOSOL-2 sont de valider les gènes candidats identifiés pour le QTL Phy-P5, et de développer un système modèle facilitant la caractérisation fonctionnelle des gènes impliqués dans l’interaction Phytophthora/Solanacées.
La variation naturelle dans les séquences des gènes candidats sera analysée chez les lignées parentales et plusieurs accessions de piment (EcoTILLING). Nous identifierons les domaines fonctionnels des gènes par analyse de recombinants intragéniques et séquençage d’allèles chimères. La validation des gènes candidats sera réalisée par transformation via Agrobacterium rhizogenes chez le piment, et par transformation stable chez la tomate. L’expression spatio-temporelle des gènes identifiés et les cascades de transduction du signal induites seront déterminées chez le piment et/ou la tomate. La validation fonctionnelle des gènes impliqués dans les réponses induites sera réalisée par génétique inverse en exploitant la plateforme TILLING de la tomate. Enfin, les résultats obtenus seront exploités pour améliorer la sélection de plantes résistantes aux Phytophthora.
Techniques utilisées par le CNRGV :
Responsable : Sonia Vautrin
- Construction de pools piment "Capsicum annum HD208" en deux dimensions
- Criblage des pools d' ADN, par PCR Quantitative en temps réel
- Séquençage des BACs Ends
- Agencement des clones BACs sur la carte physique du QTL phy-P5
Laboratoires partenaires :
-
Unité de Génétique et Amélioration des Fruits et Légumes
INRA-GAFL, UR1052
84143 Montfavet Cedex
Directeur du laboratoire : Mathilde Causse
Responsable scientifique : Véronique Lefebvre
-
Unité de Recherche en Génomique Végétale
URGV, UMR1165
INRA-CNRS-Univ
91057 Evry Cedex
Directeur du laboratoire : Michel Caboche
Responsable scientifique :Abdelhafifid Bendahmane
-
Centre National de Ressources Génomiques Végétales
INRA-CNRGV, UR1258
31326 Castanet Tolosan Cedex
Directeur du laboratoire / Responsable scientifique : Hélène Bergès

