ZeaWall : Vers l'identification de déterminants génétiques contrôlant la dégradabilité de la paroi secondaire lignifiée chez le maïs.

Coordinateurs du projet :
INRA URP3F
Yves Barrière
Le Chêne - RD 150
BP 80006
86600 Lusignan
Mail : Yves.Barriere@lusignan.inra.fr
INRA IJPB
Matthieu Reymond
Route de Saint Cyr - RD10
78026 Versailles cedex
Mail : Matthieu.Reymond@versailles.inra.fr
Partenaires :
Entreprises semencières membres de ProMaïs (Site web: http://pro-mais.org)
INRA-CNRGV
Hélène Berges
24 Chemin de Borde Rouge
31326 Castanet Tolosan
Mail : helene.berges@toulouse.inra.fr
UMR de Génétique Végétale
Clémentine Vitte
Ferme du Moulon
91190 Gif-sur-Yvette, France
Mail : vitte@moulon.inra.fr
Résumé :
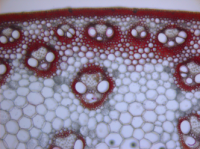
La dégradabilité des parois des graminées est un facteur limitant à la fois leur valeur énergétique pour l'alimentation des ruminants et le rendement d’hydrolyse des polysaccharides pariétaux pour la production de biocarburants de seconde génération. L’identification des déterminismes génétiques contrôlant la formation des parois lignifiées chez les graminées est donc un enjeu majeur, tant au niveau fondamental en raison des caractéristiques spécifiques des parois des monocotylédones, qu’au niveau appliqué dans l’optique d’améliorer la dégradabilité de ces dernières.
Plusieurs programmes de recherche menés en collaboration entre l’INRA et ProMaïs ont permis la détection de QTLs de dégradabilités de parois, ainsi que des QTLs de teneur en lignines et de composants phénoliques des parois chez le maïs. C’est ainsi qu’un cluster de QTLs majeur de dégradabilité et de composition des parois a été détecté chez la famille de lignées recombinantes F288xF271 au niveau d’un région génomique du maïs située sur le chromosome 6.
L’objectif des projets ZeaWall est d’identifier les déterminants génétiques responsables de la présence de ce cluster de QTLs majeurs.
Pour cela, le séquençage ciblé de cette région génomique a été entrepris pour les deux lignées parentales F288 et F271 via l’utilisation de banques BACs spécifiques.
Publications :
Courtial A, Méchin V, Reymond M, Grima-Pettenati J, Barrière Y, 2013. Colocalizations between several QTLs for cell wall degradability and composition in the F288 x F271 early maize RIL progeny raise the question of the nature of the possible underlying determinants and breeding targets for biofuel capacity. BioEnergy Research 7(1):142-156.
Courtial A, Thomas J, Reymond M, Méchin V, Grima-Pettenati J, Barrière Y, 2013. Targeted linkage map densification to improve cell wall related QTL detection and interpretation in maize. Theor Appl Genet 126: 1151-1165.
Implication du CNRGV :
Responsable: Audrey Courtial
Construction et criblage des banques BAC des lignées F288 et F271
Séquençage des BACs et analyses comparatives